Prokaryota

Empire
Prokaryota
Chatton, 1925
Domaines de rang inférieur
- Eubacteria
- Archaebacteria
Règnes de rang inférieur
reg. Bacteria
subreg. Unibacteria
phyl. Posibacteria
subphyl. Chloroflexi
subphyl. Endobacteria
subphyl. Teichobacteria
phyl. Archaebacteria
subphyl. Euryarchaeota
subphyl. Filarchaeota
subreg. Negibacteria
phyl. Hadobacteria
phyl. Cyanobacteria
infrareg. Eurybacteria
phyl. Negativicutes
phyl. Fusobacteria
infrareg. Gracilicutes
phyl. Spirochaetae
phyl. Proteobacteria
phyl. Sphingobacteria
phyl. Planctobacteria
Un procaryote (ou Prokaryota) est un être vivant dont la structure cellulaire ne comporte pas de noyau, et presque jamais d'organites membranés (la seule exception étant les thylakoïdes chez les cyanobactéries). Il s'agit (très vaguement) des microorganismes unicellulaires simples qu'on nomme informellement bactéries, et qui se divisent en archées et en eubactéries. Dans la classification du vivant en sept règnes, les procaryotes forment un taxon paraphylétique, regroupant ainsi des êtres vivants partageant une structure cellulaire similaire et simple. Thomas Cavalier-Smith propose en 1998 d'intégrer ces règnes dans les deux empires des Prokaryota et Eukaryota[1].
Selon la classification admise, ce taxon s'oppose aux eucaryotes, caractérisés par la présence d'un noyau et de multiples autres organites, cette division du vivant en deux étant considérée comme la plus fondamentale. On considère généralement que les eucaryotes se sont créés par assimilation de petits procaryotes au sein de plus grands.
Les premiers procaryotes étaient peut-être déjà présents lors de l'Éoarchéen (ère de l'Archéen), soit il y a plus de 3 600 millions d'années. L'étude des procaryotes s'est surtout développée au XIXe siècle, avec les travaux de Louis Pasteur en France et de Robert Koch en Allemagne. Le terme « procaryote » trouve toute sa signification dans les années 1950, lorsque le microscope électronique montre l'absence de noyau vrai dans la cellule.
Sommaire
1 Histoire et étymologie
1.1 Origine du concept
1.2 Histoire des concepts de monère, de bactérie, de procaryote
1.3 Pertinence actuelle du concept
2 Identification et classification
2.1 Classification phénotypique
2.2 Chimiotaxonomie
2.3 Classification moléculaire
2.4 Identification des espèces bactériennes
2.5 Relations avec les eucaryotes
3 Plan d'organisation
3.1 Paroi cellulaire
3.2 Génétique
3.2.1 Matériel génétique
3.2.2 Variation génétique
4 Croissance et reproduction
4.1 Division cellulaire
4.2 Croissance et culture des bactéries
4.3 Paramètres influant sur la croissance microbienne
4.4 Endospores
4.5 Métabolisme
5 Notes et références
6 Voir aussi
6.1 Bibliographie
6.2 Articles connexes
6.3 Liens externes
Histoire et étymologie |
Origine du concept |
Le terme procaryote provient du latin pro, « avant », et du grec karyon, « noyau ».
Le concept de monère (du grec moneres, « simple ») conçu par Haeckel est assez semblable.
Les procaryotes sont également désignés par Monera, Prokaryota (voire Schizophyta ou Schizomycetes).
Le terme est aussi écrit sous l'orthographe Prokarya pour les biologistes Margulis et Chapman (2009)[2], qui considèrent le taxon comme un super-règne.
Histoire des concepts de monère, de bactérie, de procaryote |
Leeuwenhoek fut le premier à observer des bactéries, grâce à un microscope de sa fabrication, en 1668[3]. Il les appela « animalcules » et publia ses observations dans une série de lettres qu'il envoya à la Royal Society[4],[5],[6].
Le mot « bactérie » apparaît pour la première fois avec le microbiologiste allemand Ehrenberg en 1838. Ehrenberg classait les bactéries en tant que vibrions dans le règne animal. En 1857, Nägeli les plaçait parmi les Plantes, dans le groupe des Schizomycetes[7]. Parallèlement Haeckel inventa, en 1866[8], l'embranchement Monera pour regrouper, au sein de son règne Protista, tous les micro-organismes sans structure interne (bien qu'excluant les cyanobactéries, alors classées parmi les végétaux). Cohn utilisa à son tour le terme Bacteria comme taxon en 1870 et tenta le premier de les classer rigoureusement selon leur morphologie. Pour Cohn, les bactéries étaient des plantes primitives non chlorophylliennes. Cohn les classa en tant que végétaux inférieurs dans l'embranchement des Schizophytes. À la suite des travaux de Cohn, Haeckel révisa la circonscription de ses "monères" pour y inclure les cyanobactéries[9]. Les termes de "monère" et de "bactérie" devinrent alors synonymes.
En 1925[10], dans un essai de classification des Protistes, Chatton forge le groupe taxinomique des Procaryotes constitué des Cyanophycae (Cyanophycées), des Bacteriacae (Bactériacées) et des Spirochaetacae (Spirochétacées) ainsi que celui des Eucaryotes formé des protozoaires Mastigiae (Flagellés, Rhizopodes et Sporozoaires), Ciliae (Ciliés) et Cnidiae (Cnidosporidies et Acnidosporidies).
En 1937[11], Chatton propose une classification du monde du vivant en deux types cellulaires qu'il nomme Procaryotes (organismes à cellules sans noyau) et Eucaryotes (organismes à cellules avec noyau). La notion de Procaryotes recouvre alors celle de Protistes inférieurs.
En 1938, Copeland éleva les monères au rang de règne, à un niveau désormais égal aux animaux, plantes et protistes[12].
Traçant les grandes lignes de la classification bactérienne en 1941, Stanier et Van Niel proposèrent de subdiviser le règne Monera en deux divisions : I. Myxophyta pour les algues bleues et II. Schizomycetae pour les bactéries[13].
En 1957, Dougherty classa les organismes vivants en deux grands groupes, dont il nomma formellement les structures nucléiques prokaryon et eukaryon (au singulier), prokarya et eukarya (au pluriel), sur la base de l'absence ou de la présence d'un noyau bien défini (délimité par une membrane nucléaire)[14] et plaça les bactéries avec les algues bleues dans le premier groupe Monera[15].
Ce n'est qu'en 1957 que Lwoff distingua avec clarté les concepts de bactérie et de virus[16] grâce à des arguments biochimiques et structuraux.
En 1961, Stanier adopte la terminologie proposée par Chatton en désignant deux types de cellules : « La cellule du type qui existe chez les bactéries et les algues bleues est une cellule procaryote ; la cellule du type qui existe chez les autres organismes, est une cellule eucaryote[17]. »
Enfin Stanier et Van Niel définirent pour la première fois rigoureusement, en 1962, le concept de bactérie par l’absence d’organite membrané (et en particulier de véritable noyau, donc de mitose) et réintroduisirent le terme de « procaryote » auprès de la communauté scientifique internationale[18].
Murray créa le règne des procaryotes, en 1968, sous le nom formel de Procaryotae[19],[20].
Allsopp proposa, en 1969, que le règne comprenant les Bacteria et les Cyanophyta soit nommé Procaryota[21].
Pertinence actuelle du concept |
Les procaryotes forment un taxon paraphylétique, sa pertinence a donc été remise en cause par l'école cladiste et notamment Carl Woese[22].
Cependant, l'existence d'un plan d'organisation commun aux archébactéries et aux eubactéries rend la reconnaissance de ce groupe indispensable pour l'école évolutionniste[23].
Identification et classification |
La taxonomie permet de classer de façon rationnelle les organismes vivants. Chez les bactéries, les taxons dans l’ordre hiérarchique sont les suivants : phylums (ou divisions), classes, sous-classes, ordres, sous-ordres, familles, sous-familles, tribus, sous-tribus, genres, sous-genres, espèces et sous-espèces. Différentes approches permettent la classification des bactéries.
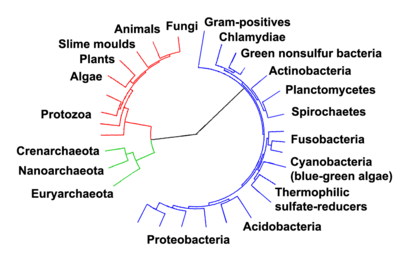
Arbre phylogénétique montrant la diversité des bactéries, comparés aux autres organismes[24]. Les eucaryotes sont colorés en rouge, les archaea en vert et les bactéries en bleu.
Classification phénotypique |
- Critères morphologiques (forme et groupement des bactéries, présence ou absence de flagelle, nature de la paroi, type de mobilité, présence d’endospore).
- Critères physiologiques (type métabolique, source d’énergie, de carbone, d’azote, type de substrat utilisé, capacité à produire certaines molécules, produits de fermentation, métabolites secondaires, etc.).
- Critères de pathogénicité.
- Critères de sérogroupage.
Chimiotaxonomie |
Il s’agit de l’analyse chimique de constituants cellulaires (structure et composition de la paroi, des membranes plasmiques, du peptidoglycane).
Classification moléculaire |
- Aujourd'hui (2013), la phylogénie moléculaire des bactéries se base de plus en plus sur la comparaison de différentes espèces à l'échelle du génome grâce aux progrès du séquençage.
- Les séquences des gènes codant les ARN ribosomiques (ARNr) peuvent être utilisés soit pour établir une phylogénie moléculaire des bactéries dans leur ensemble, soit pour identifier très précisément la taxonomie d'une bactérie (voir barcoding moléculaire), selon que les parties conservées ou plus variables de ces séquences sont considérées.
- Le Multilocus Sequence Typing (MLST, «Typage moléculaire multilocus») est une approche semblable au séquençage des ARNr mais s'appuyant sur plusieurs gènes de ménage.
- L'hybridation ADN-ADN est une technique qui permet d'estimer la similarité globale entre les génomes de deux bactéries proches. Cette mesure entre parfois dans la définition des espèces bactériennes. Néanmoins, ces comparaisons tendent à être de moins en moins utilisés du fait l'avènement de la génomique, qui permet de travailler avec les génomes eux-mêmes.
Identification des espèces bactériennes |
La détermination génétique des espèces se base sur l’étude des gènes des ARN ribosomiques. Le choix des gènes des ARNr 16S se justifie pour les raisons suivantes :
- les ARNr 16S sont des molécules ubiquistes ;
- leur structure est bien conservée car toute modification pourrait nuire à la synthèse protéique. Il en résulte une évolution très lente de ces gènes.
Le choix des gènes des ARNr plutôt que les ARNr eux-mêmes se base sur le choix de la technique de l’amplification par PCR. Cette technique permet, à partir d’une colonie de bactéries, d’obtenir des fragments d’ADN correspondants au gène ou à une partie du gène.
Les analyses génétiques concernent également la région intergénique 16S-23S des opérons des ARN ribosomiques. Cette dernière est une région de longueur variable selon les organismes. Elle donne une indication immédiate sur le fait que deux souches données appartiennent ou non à la même espèce.
Tous les micro-organismes possèdent au moins une copie des gènes codant les ARN ribosomiques. Ces molécules sont indispensables à la synthèse des protéines, raison pour laquelle cette séquence d’ADN est très conservée au sein des espèces (plus de 99 %). Cette conservation de séquence permet d’utiliser cette région pour la détermination des espèces. En effet, Le degré de similitude des séquences d’ARNr entre deux organismes indique leur parenté relative. La procédure utilisant l’ARNr 16S comme facteur d'identification implique l'extraction de l’ADN des bactéries d’une colonie. Puis des amorces reconnaissant des zones très conservées du gène permettent d'amplifier par PCR une grande partie du gène ARNr 16S, qui par la suite est séquencé. Les données sur la séquence nucléotidique sont comparées avec des bases de données de séquences déjà connues.
Les séquences du gène codant l’ARNr 16S sont connues pour plus de 4 000 souches bactériennes. Ces séquences peuvent être consultées par interrogation de banques de données (fichiers au formats EMBL, GenBank, Fasta) par des logiciels comme le Blast. Le Ribosomal Data Project II (RDP) est également intéressant dans la mesure où sa base de données est spécifique de l’ARN 16S. Ces logiciels sont accessibles en ligne sur l’internet. Selon les différents auteurs, le degré d’homologie entre deux bactéries pour qu’elles appartiennent à la même espèce doit être supérieur à 97 %, voire 99 %.
Comme les gènes de la région intergénique 16S-23S sont moins conservés, ils diffèrent d’une souche à l’autre aussi bien au niveau de la séquence que de la longueur. Ceci résulte de ce que de nombreuses bactéries ont des copies multiples par génome de l’opéron de l’ARNr, il en résulte lors de l’amplification un motif caractéristique. Comme pour le gène de l’ARNr 16S, l’étude systématique de la région intergénique 16S-23S requiert l’amplification de cette région par PCR. L’utilité de la région intergénique 16S-23S est qu’elle permet de distinguer des espèces différentes et parfois différentes souches au sein de la même espèce. En effet la région intergénique étant moins conservée, des variabilités au niveau des séquences peuvent se présenter pour des souches de la même espèce mais appartenant à des biovars différents.
Les séquences de la région intergénique 16S-23S sont comparées par interrogation des bases de données IWoCS qui est spécifique de la région intergénique 16S-23S. La base de données GenBank est également très bien fournie. Le degré d’homologies devrait idéalement être proche de 100 % pour des souches identiques.
Relations avec les eucaryotes |
La distinction principale entre les procaryotes et les eucaryotes se base sur le fait que le matériel génétique des procaryotes est regroupé dans une zone appelée nucléoïde qui n'est pas physiquement séparée du reste de la cellule, alors que chez les eucaryotes, celui-ci est contenu par un organite, le noyau[25]. Les organismes eucaryotes peuvent être unicellulaires, comme les amibes, ou pluricellulaires, comme les plantes et les humains. La différence entre la structure des procaryotes et des eucaryotes est si grande qu'elle est parfois considérée comme la distinction la plus importante entre tous les groupes d'organismes. Toutefois, une critique de cette classification est que le mot procaryote est basé sur ce que ces organismes ne sont pas (eucaryotes), plutôt que ce qu'ils sont (archées ou bactéries)[26]. En 1977, Carl Woese propose de diviser les procaryotes entre les bactéries et les archaea (à l'origine des eubactéries et des archaebactéries) en raison des grandes différences au niveau de la structure et de la génétique des deux groupes d'organismes.

« Anneau » de la vie : forme circulaire d'arbre phylogénétique des Procaryotes montrant l'origine des Eucaryotes selon la théorie d'une symbiose entre Procaryotes aérobies et anaérobies.
Selon la théorie endosymbiotique énoncée par Lynn Margulis (1967) puis par Max Taylor (1974)[27], les cellules Eucaryotes proviennent de l'association de plusieurs Procaryotes.
Admettant la théorie de la fusion des génomes entre Bactéries et Archées (pour engendrer les Eucaryotes), d'autres biologistes comme Rivera et Lake (2004)[28] tendent à remplacer l'image de l'arbre phylogénétique des Procaryotes par une forme circulaire, ou « anneau » de la vie
[29],[30].
Le texte qui suit est une comparaison des caractéristiques des Procaryotes et des Eucaryotes:
Procaryotes
| Eucaryotes
|
Plan d'organisation |

structure typique d'une cellule procaryote.
 Lire le média
Lire le médiaAnimation 3D d'une cellule procaryote qui montre tous les éléments qui le composent.
Les monères sont des bactéries et archées dont la taille est de l'ordre de 0,01 mm, comme les coques, les bacilles ou les spirilles. Les procaryotes possèdent une paroi cellulaire (polypeptides, polysaccharides) et un ADN circulaire généralement unique (de nombreux procaryotes ont plusieurs chromosomes comme Rhodobacter qui en possède deux ou Deinococcus qui en a quatre). Cet ADN est associé aux protéines HU et IHF. Les procaryotes possèdent également parfois des plasmides. À l'inverse du noyau chez les cellules eucaryotes, la cellule procaryote possède un filament d'ADN qui contient l'information génétique qui n'est protégée que par une membrane nucléaire.
Les procaryotes étaient vues comme de simples sacs de cytosol, mais de nombreux niveaux de complexité structurelle ont été découverts depuis, comme la découverte du cytosquelette procaryote[33],[34], et la localisation spécifique de protéines dans le cytoplasme bactérien[33]. Ces compartiments subcellulaires ont été nommés « hyperstructure bactériennes » (« bacterial hyperstructures » en anglais)[35].
Les bactéries possèdent un chromosome sous forme de filament d’ADN, support de l’hérédité. Le chromosome bactérien est en général unique et circulaire. En plus de cet ADN génomique, les cellules bactériennes contiennent souvent des molécules d’ADN circulaire extra-chromosomiques appelées plasmides. Les cellules contiennent aussi de nombreux ribosomes permettant la synthèse protéique grâce au mécanisme de la traduction. Le cytosol des procaryotes contient souvent des substances intracellulaires de réserve qui sont des stocks de nutriments sous forme de glycogène, amidon ou poly-b-hydroxybutyrate (PBH). Certaines espèces de bactéries aquatiques possèdent des vésicules à gaz qui assurent la flottabilité des cellules. D’autres espèces, les bactéries magnétotactiques, ont la particularité de présenter un magnétosome.
Paroi cellulaire |
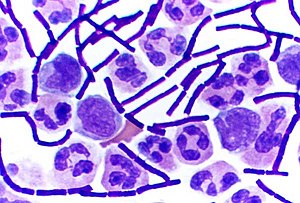
Bacillus anthracis (bâtonnets violets foncé, donc à Gram positif) se développant dans du liquide céphalorachidien.
Une caractéristique importante des bactéries est la paroi cellulaire. La paroi donne à la bactérie sa forme et la protège contre l’éclatement sous l’effet de la très forte pression osmotique du cytosol. Les bactéries peuvent être structuralement divisées en deux groupes : les bactéries à paroi unimembranée (ne contenant qu'une seule membrane, la membrane plasmique) et les bactéries à paroi bimembranée (constituée de deux membranes superposées, la membrane interne et la membrane externe). Les premières peuvent être appelées Unibacteria et les secondes Negibacteria. Cette structure peut être révélée par un simple test chimique, la coloration de Gram. Les unibactéries sont généralement à Gram positif alors que les négibactéries sont à Gram négatif.
La rétention du colorant par les bactéries à Gram positif est due à la possession d'un peptidoglycane (ou muréine) épais sur la face externe de la membrane plasmique, alors que les bactéries à Gram négatif présentent généralement un peptidoglycane fin localisé dans le périplasme entre les deux membranes de la paroi. Cependant ce critère empirique n'est pas parfait. En effet certaines unibactéries comme les mycoplasmes sont à Gram négatif à cause de l'absence de peptidoglycane dans leur paroi. De même les archébactéries, qui ont une structure unibactérienne, peuvent être à Gram négatif ou à Gram positif selon la composition et l'épaisseur de leur paroi (qui est le plus souvent un pseudopeptidoglycane). Certaines négibactéries sont également complètement dépourvues de peptidoglycane comme les planctobactéries.
Génétique |
Matériel génétique |
La plupart des bactéries possèdent un unique chromosome circulaire. Il existe toutefois de rares exemples de bactéries, comme Rhodobacter sphaeroides possédant deux chromosomes. Les bactéries du genre Borrelia ont la particularité d'avoir un génome linéaire et segmenté, ce qui est exceptionnel chez les procaryotes. La taille du génome peut être très variable selon les espèces de bactéries étudiées.
Le génome de la souche de Escherichia coli séquencé en 1997 est constitué de 4,6 Mpb (4 600 000 paires de bases), il code 4 200 protéines.
Le génome d’une autre souche de E. coli séquencé en 2001 comprend 5,5 Mpb codant 5 400 protéines.
Certaines bactéries présentent un tout petit génome, comme la bactérie parasite Mycoplasma genitalium avec un génome de 580 000 paires de bases et la bactérie endosymbiotique d’insecte, Candidatus Carsonella ruddii avec un génome de seulement 160 000 paires de bases[36].
Au contraire, la bactérie du sol Sorangium cellulosum possède un génome constitué de 12 200 000 paires de bases[37].
Chose peu commune, les Spirochètes ainsi que des Streptomyces présentent la particularité d’avoir un chromosome linéaire[38].
Les bactéries contiennent également souvent un ou plusieurs plasmides, qui sont des molécules d’ADN extra-chromosomique.
Ces plasmides peuvent conférer certains avantages aux bactéries, comme la résistance à des antibiotiques ou des facteurs de virulence. Les plasmides sont généralement des ADN double brin circulaire. Ils se répliquent indépendamment du chromosome.
Le chromosome bactérien peut d’autre part intégrer de l’ADN de virus bactérien (bactériophage).
Ces bactériophages peuvent contribuer au phénotype de l’hôte[39].
Par exemple, les bactéries Clostridium botulinum et Escherichia coli O157:H7 synthétisent une toxine codée par un gène qui provient d’un phage qui s’est intégré au génome de ces bactéries au cours de l’évolution[40].
Variation génétique |
Les bactéries sont des organismes asexués ; après la division bactérienne, les cellules filles héritent d’une copie identique du génome de leur parent. Cependant, toutes les bactéries sont capables d’évoluer par modification de leur matériel génétique causé par des recombinaisons génétiques ou des mutations. Les mutations (changements plus ou moins ponctuels et aléatoires de l'information génétique d'une cellule) proviennent d’erreur durant la réplication de l’ADN ou de l’exposition à des agents mutagènes. Le taux de mutation varie grandement selon les espèces ou les souches bactériennes[41].
Quelques bactéries peuvent également transférer du matériel génétique entre les cellules. Il existe trois mécanismes de transfert de gènes entre les cellules : la transformation, la transduction, et la conjugaison.
Au cours de la transformation, c’est un plasmide qui est transféré dans la cellule bactérienne, alors qu’au cours de la transduction, le transfert d’ADN a lieu par l’intermédiaire d’un bactériophage. Au cours de la conjugaison, deux bactéries peuvent se rapprocher, grâce à des structures spéciales, les pili, et il y a alors un transfert d’ADN d’une bactérie à une autre. L’ADN étranger peut être intégré dans le génome et être transmis aux générations suivantes. Cette acquisition de gènes, provenant d’une bactérie ou de l’environnement, est appelé transfert horizontal de gènes (HGT pour horizontal gene transfer)[42]. Le transfert de gènes est particulièrement important dans les mécanismes de résistance aux antibiotiques[43].
Croissance et reproduction |
Division cellulaire |
Les archébactéries et les eubactéries se multiplient par scissiparité, ou parfois par gemmiparité. Bien que des mécanismes modifiant le génome des procaryotes existent (comme des mutations, recombinaisons ou encore des transferts horizontaux de gènes), on ne parle pas de reproduction.
Deux cellules identiques sont produites à partir d’une cellule mère. La croissance cellulaire se manifeste par un accroissement du volume cellulaire, suivi de la synthèse d’un septum transversal au milieu de la cellule, aboutissant à la séparation des deux cellules filles. La division bactérienne est précédée par la duplication du chromosome bactérien grâce à la réplication de l'ADN. De nombreuses protéines interviennent durant ces processus, notamment des protéines considérées comme des équivalents de cytosquelette bactériens tels que la protéine ATPase FtsZ.
Quelques bactéries présentent des structures reproductives plus complexes mais toujours de manière asexuée, facilitant la dispersion : Myxococcus élabore des fructifications (corps fructifiants), tandis que Streptomyces forme des hyphes aériens.
Quand elles se trouvent dans un milieu propice, les bactéries peuvent se multiplier à une allure vertigineuse. Une population de bactéries peut doubler toutes les 20 minutes en fonction de : la disponibilité en nutriments, la présence de bactéries concurrentes, la présence de prédateurs (par exemple des paramécies), la présence de bactériophages, la présence d’antibiotiques (inhibant par exemple la synthèse de la paroi bactérienne, entraînant donc leur mort) produits par des champignons ou des actinomycètes (bactéries filamenteuses).
Croissance et culture des bactéries |

Colonies bactériennes sur milieu solide gélosé en boîte de Pétri.

Biofilm bactérien, près d'une source chaude du Parc de Yellowstone

Les bactéries sont responsables de la remontée de bulles de méthane et de flocs bactériens, à partir des sédiments anoxique et très riches en matière organique (ici sur la Basse-Deûle un jour férié sans circulation de péniches)
Dans la nature, depuis des milliards d'années, les biofilms et concrétions bactériennes contribuent au cycle de nombreux éléments, à la formation de « filons » riches en métaux (par fixation en biofilm et/ou bioconcentration[44]), ainsi qu'à la formation et dégradation des roches.
Au laboratoire, les bactéries peuvent être cultivées en milieu de culture liquide ou en milieu solide. Le milieu de culture doit apporter les éléments nutritifs ou nutriments élémentaires à la bactérie. Les milieux de culture gélosés solides sont utilisés pour isoler des cultures pures de cellules bactériennes. Dans le cas des bactéries se divisant rapidement, une cellule bactérienne dispersée sur un milieu gélosé va se multiplier et, au bout de 24 à 48 heures, devenir un amas de bactéries, appelé une colonie bactérienne, visible à l’œil nu.
Le temps de génération est le temps nécessaire à une bactérie pour se diviser. Le temps de génération correspond donc au temps nécessaire pour qu’une population de cellules double en nombre. Ce temps est très variable selon les espèces de bactéries et les conditions environnementales. Au laboratoire, dans des conditions idéales, il est par exemple de 20 minutes pour Escherichia coli, 100 minutes pour Lactobacillus acidophilus, 1 000 minutes pour Mycobacterium tuberculosis.
La croissance d’une population bactérienne dans un milieu de culture liquide non renouvelé, peut être observée dans le temps. Les cellules se divisent, et leur nombre augmente avec le temps. Si on relève le nombre de bactéries à différents intervalles au cours de la croissance, on obtient une courbe de croissance. Elle présente quatre phases principales :
- La phase de latence correspond à une période d’adaptation de la bactérie au milieu
- Au cours de la phase de croissance exponentielle, les bactéries se développent de façon maximale avec un taux de croissance maximal et constant
- Après une phase transitoire de ralentissement, le nombre de bactéries n’évolue plus : c’est la phase stationnaire. Les divisions bactériennes qui se font encore sont compensées par la mort de bactéries
- La dernière phase est la phase de mortalité ou de déclin. Les bactéries ne se divisent plus, elles meurent et peuvent être lysées. Le milieu de culture n’apporte plus les conditions nécessaires au développement des bactéries. On observe une courbe de décroissance exponentielle progressive.
Paramètres influant sur la croissance microbienne |
Certaines conditions environnementales (paramètres physico-chimiques) influencent la croissance des micro-organismes. Parmi celles-ci figurent le pH (acidité et alcalinité), la température, la présence d’O2, de CO2, la disponibilité de l’eau.
La plupart des micro-organismes tolèrent une gamme de pH permettant la croissance. Le pH optimal de croissance de beaucoup de bactéries est proche de la neutralité (pH 7). Les micro-organismes acidophiles se développent à des pH acides, alors que les micro-organismes alcalinophiles se développent à des pH basiques.
De même, les bactéries peuvent être distinguées selon leur aptitude à croître en fonction de la température. Les mésophiles se développent généralement à des températures comprises entre 20 et 45 °C. Les psychrophiles possèdent des températures optimales de croissance inférieures à 15 °C, alors que les bactéries thermophiles croissent de façon optimale à des températures comprises entre 45 et 70 °C. Les micro-organismes ayant des températures optimales de croissance supérieures à 70 °C sont qualifiés d’hyperthermophiles.
Endospores |
Quelques bactéries, Gram positif, comme Bacillus, Clostridium, Sporohalobacter, Anaerobacter et Heliobacterium peuvent fabriquer des endospores leur permettant de résister à certaines conditions de stress environnemental ou chimique[45].
La formation d'un endospore n'est pas un processus de reproduction. Les Anaerobacter peuvent faire jusqu'à sept endospores en une seule cellule[46]. Les bactéries à endospores ont une zone centrale de cytoplasme contenant l'ADN et ribosomes entouré par une couche du cortex et protégé par un manteau imperméable et rigide.
Les bactéries à endospores peuvent survivre dans des conditions physiques et chimiques extrêmes, tels que des niveaux élevés de rayonnement UV, les rayons gamma, les détergents, les désinfectants, une forte chaleur ou pression et à la dessiccation[47]. Ces organismes pourraient rester viable durant des millions d'années[48],[49]. Les endospores peuvent même permettre aux bactéries de survivre à l'exposition au vide et au rayonnement dans l'espace[50].
Les bactéries à endospore peuvent également causer des maladies: par exemple, la maladie du charbon peut être contractée par l'inhalation d'endospores Bacillus anthracis, et la contamination des plaies profondes avec la perforation par Clostridium tetani responsable du tétanos[51].
Métabolisme |
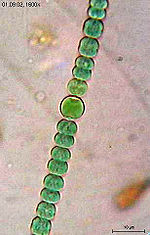
Une cyanobactérie : Anabaena sperica.
Le métabolisme d’une cellule est l’ensemble des réactions chimiques qui se produisent au niveau de cette cellule. Pour réaliser ce processus, les bactéries, comme toutes les autres cellules, ont besoin d’énergie. L’ATP est la source d’énergie biochimique universelle. L’ATP est commune à toutes les formes de vies, mais les réactions d’oxydo-réduction impliquées dans sa synthèse sont très variées selon les organismes et notamment chez les bactéries. Les bactéries vivent dans pratiquement toutes les niches environnementales de la biosphère. Elles peuvent ainsi utiliser une très large variété de source de carbone et/ou d’énergie.
Les bactéries peuvent être classées selon leur type de métabolisme, en fonction des sources de carbone et d’énergie utilisés pour la croissance, les donneurs d’électrons et les accepteurs d’électrons.
L’énergie cellulaire des chimiotrophes est d’origine chimique alors que celle des phototrophes est d’origine lumineuse. La source de carbone des autotrophes est le CO2, tandis que des substrats organiques sont la source de carbone des hétérotrophes. Il est aussi possible de distinguer deux sources possibles de protons (H+) et d'électrons (e-) : les bactéries réduisant des composés minéraux sont des lithotrophes alors que celles réduisant des substances organiques sont des organotrophes.
Les bactéries peuvent être divisées en quatre grands types nutritionnels en fonction de leurs sources de carbone et d’énergie :
- Les photoautotrophes utilisent la lumière comme source d’énergie et le CO2 comme source de carbone.
- Les photohétérotrophes se développent par photosynthèse. Ils assimilent le CO2 en présence d’un donneur d’électrons.
- Les chimioautotrophes utilisent des substrats inorganiques réduits pour l’assimilation réductrice du CO2 et comme source d’énergie.
- Les chimiohétérotrophes utilisent des substrats organiques comme source de carbone et d’énergie.
Chez les chimiohétérotrophes, les substrats sont dégradés en plus petites molécules pour donner des métabolites intermédiaires (pyruvate, acétylCoA…) qui sont eux-mêmes dégradés avec production de CO2, H2O et d’énergie. Ces réactions productrices d’énergie sont des réactions d’oxydation d’un substrat hydrogéné, avec libération de protons et d’électrons grâce à des déshydrogénases. Le transfert de protons et d’électrons à un accepteur final est réalisé par toute une série d’enzymes qui forment une chaîne de transport électronique. L’énergie ainsi produite est libérée par petites étapes dans le but d’être transférée dans des liaisons chimiques riches en énergie (ATP, NADH, NADPH). Suivant la nature de l’accepteur final d’électrons, on distingue les processus de la respiration et de la fermentation. La respiration peut être aérobie quand O2 est l’accepteur final de protons et d’électrons, ou anaérobie (respiration nitrate, et respiration fumarate par exemple). Dans tous les cas, l’accepteur final d’électrons doit être une molécule oxydée (O2, NO3−, SO2−).
Chez les organismes aérobies, l’oxygène est utilisé comme accepteur d’électrons. Chez les organismes anaérobies, d’autres composés inorganiques comme le nitrate, le sulfate ou le dioxyde de carbone sont utilisés comme accepteurs d’électrons. Ces organismes participent à des processus écologiques très importants lors de la dénitrification, la réduction des sulfates et l’acétogenèse. Ces processus sont aussi importants lors de réponses biologiques à la pollution, par exemple, les bactéries réduisant les sulfates sont responsables de la production de composés hautement toxiques à partir du mercure (méthyl et diméthylmercure) présent dans l’environnement.
Les anaérobies (non respiratoires) utilisent la fermentation pour fournir de l’énergie à la croissance des bactéries. Au cours de la fermentation, un composé organique (le substrat ou la source d’énergie) est le donneur d’électrons tandis qu’un autre composé organique est l’accepteur d’électrons. Les principaux substrats utilisés lors de la fermentation sont des glucides, des acides aminés, des purines et des pyrimidines. Divers composés peuvent être relargués par les bactéries lors des fermentations. Par exemple, la fermentation alcoolique conduit à la formation d’éthanol et de CO2. Les bactéries anaérobies facultatives sont capables de modifier leur métabolisme entre la fermentation et différents accepteurs terminaux d’électrons, selon les conditions du milieu où elles se trouvent.
Selon leur mode de vie, les bactéries peuvent être classées en différents groupes :
- Les aérobies strictes peuvent vivre uniquement en présence de dioxygène ou oxygène moléculaire (O2).
- Les aéro-anaérobies facultatives peuvent vivre en présence ou en absence de dioxygène.
- Les anaérobies ne peuvent vivre qu'en absence de dioxygène. Les aérotolérants sont des organismes anaérobies qui peuvent tout de même survivre en présence d’oxygène.
- les microaérophiles requièrent de l’oxygène pour survivre mais à une concentration faible.
Les bactéries lithotrophes peuvent utiliser des composés inorganiques comme source d’énergie. L’hydrogène, le monoxyde de carbone, l’ammoniac (NH3), les ions ferreux ainsi que d’autres ions métalliques réduits et quelques composés du soufre réduit. Le méthane peut être utilisé par les méthanotrophes comme source de carbone et d’électrons. Chez les phototrophes aérobie et les chimiolithotrophe, l’oxygène est utilisé comme accepteur terminal d’électrons, alors qu’en condition anaérobie, ce sont des composés inorganiques qui sont utilisés.
En plus de la fixation du CO2 lors de la photosynthèse, quelques bactéries peuvent fixer l’azote N2 (fixation de l’azote) en utilisant une enzyme : la nitrogénase. Des bactéries aérobies, anaérobies et photosynthétiques sont capables de fixer l’azote. Les cyanobactéries qui fixent l’azote, possèdent des cellules spécialisées (les hétérocystes).
Notes et références |
(en) Thomas Cavalier-Smith, 1998. A revised six-kingdom system of life. Biol. Rev. 73: 203-266.
(en) Lynn Margulis & Michael J. Chapman, Kingdoms and Domains : An Illustrated Guide to the Phyla of Life on Earth, Academic Press, Boston, 2009, 731 p. (ISBN 978-0-12-373621-5)
(en) J.R. Porter, « Antony van Leeuwenhoek: Tercentenary of his discovery of bacteria », Bacteriological reviews, vol. 40, no 2, juin 1976, p. 260-269 (PMID 786250, PMCID 413956, lire en ligne [PDF])
(en) A. van Leeuwenhoek, « An abstract of a letter from Mr. Anthony Leevvenhoek at Delft, dated Sep. 17, 1683, Containing Some Microscopical Observations, about Animals in the Scurf of the Teeth, the Substance Call'd Worms in the Nose, the Cuticula Consisting of Scales », Philosophical Transactions (1683–1775), vol. 14, 1684, p. 568–574 (DOI 10.1098/rstl.1684.0030, lire en ligne [PDF])
(en) A. van Leeuwenhoek, « Part of a Letter from Mr Antony van Leeuwenhoek, concerning the Worms in Sheeps Livers, Gnats, and Animalcula in the Excrements of Frogs », Philosophical Transactions (1683–1775), vol. 22, 1700, p. 509–518 (DOI 10.1098/rstl.1700.0013, lire en ligne[archive du 25 mars 2016] [PDF])
(en) A. van Leeuwenhoek, « Part of a Letter from Mr Antony van Leeuwenhoek, F. R. S. concerning Green Weeds Growing in Water, and Some Animalcula Found about Them », Philosophical Transactions (1683-1775), vol. 23, 1702, p. 1304–11 (DOI 10.1098/rstl.1702.0042, lire en ligne [PDF])
(de) Carl von Nägeli, "Über die neue Krankheit der Seidenraupe und verwandte Organismen", Botanische Zeitung, 1857, Vol.15, p.760-761. [lire en ligne]
(de) E. Haeckel, Generelle Morphologie der Organismen, Reimer, Berlin, 1866.
(en) E. Haeckel, Wonders of life, Watts, London, 1904, 501 p.
É. Chatton, « Pansporella perplexa, amœbien à spores protégées parasite de daphnies : Réflexions sur la biologie et la phylogénie des protozoaires », Annales des Sciences Naturelles : Zoologie concernant l'anatomie, la physiologie, la classification et l'histoire naturelle des Animaux, Dixième série, Tome VIII, N°1 et 2, Avril 1925, p.5-84. disponible sur Gallica
É. Chatton, Titres et Travaux Scientifiques (1906–1937), Sottano, Sète (France), 1937.
(en) H.F. Copeland, "The Kingdoms of Organisms", The Quarterly Review of Biology, Vol.13, No.4, December 1938, p.383-420 DOI:10.1086/394568
(en) R.Y. Stanier & C.B. van Niel, "The Main Outlines of Bacterial Classification", Journal of Bacteriology, October 1941, Vol.42, No.4, p.437-466. PMC 374769
(en) E.C. Dougherty, "Neologisms needed for structures of primitive organisms : 1. Types of nuclei", The Journal of Protozoology, Vol.4, No.S3 (Supplement), August 1957, Abstract 55, p.14. DOI:10.1111/j.1550-7408.1957.tb02521.x
(en) E.C. Dougherty, "A Revised Classification for the Higher Taxa of the Monera", The Journal of Protozoology, Vol.4, No.S3 (Supplement), August 1957, Abstract 57, p.14. DOI:10.1111/j.1550-7408.1957.tb02521.x
(en) A. Lwoff, "The concept of virus", Journal of General Microbiology, Vo.17, No.2, October 1957, p.239-253. DOI:10.1099/00221287-17-2-239
R.Y. Stanier, « La place des bactéries dans le monde vivant », Annales de l'Institut Pasteur, Tome 101, N°3, Septembre 1961, p.297-312. disponible sur Gallica
(en) R.Y. Stanier & C.B. van Niel, "The concept of a bacterium", Archiv für Mikrobiologie, Vol.42, No.1, March 1962, p.17-35. DOI:10.1007/BF00425185
(en) R.G.E. Murray, "Microbial structure as an aid to microbial classification and taxonomy", Spisy Fac. Sci. Univ. Purkyne (Brno), Vol.43, 1968, p.245-252.
Alain Branger, Marie-Madeleine Richer et Sébastien Roustel, Alimentation, sécurité et contrôles microbiologiques, Educagri éditions, Dijon, 2007, p.17. (ISBN 978-2-84444-616-9)
(en) Allan Allsopp, « Phylogenetic relationships of the Procaryota and the origin of the eucayotic cell », New Phytologist, vol. 68, no 3, juillet 1969, p. 591-612 (ISSN 0028-646X, DOI 10.1111/j.1469-8137.1969.tb06464.x).
(en) Carl R. Woese, "Default taxonomy : Ernst Mayr’s view of the microbial world", Proc. Natl. Acad. Sci. USA, Vol.95, September 15, 1998, p.11043–11046. DOI:10.1073/pnas.95.19.11043
(en) William Martin & Eugene V. Koonin (2006). A positive definition of prokaryotes. Nature 442, 868
(en) Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P, « Toward automatic reconstruction of a highly resolved tree of life », Science, vol. 311, no 5765, 2006, p. 1283–7 (PMID 16513982, DOI 10.1126/science.1123061)
Lansing M. Prescott, John P. Harley et Donald A. Klein (trad. Claire-Michèle Bacq-Calberg, Jean Dusart), Microbiologie, De Boeck (lire en ligne)
(en) Sapp J, « The prokaryote-eukaryote dichotomy: meanings and mythology », Microbiol. Mol. Biol. Rev., vol. 69, no 2, juin 2005, p. 292–305 (PMID 15944457, PMCID 1197417, DOI 10.1128/MMBR.69.2.292-305.2005, lire en ligne)
(en) Taylor, F.J.R. 1974, Implications and extensions of the Serial Endosymbiosis Theory of the origin of eukaryotes Taxon 23 (2/3): 229-258.
(en) Maria C. Rivera & James A. Lake (2004), "The ring of life provides evidence for a genome fusion origin of eukaryotes", Nature 431 (7005) : 152-155, 9 September 2004. DOI:10.1038/nature02848
Jacques van Helden, « Chapitre 9 : Ce que nous apprend l'analyse des génomes au sujet de l'évolution », sous la direction de Gérard Cobut, Comprendre l'évolution 150 ans après Darwin, coll. « Action ! », De Boeck, Bruxelles, 2009, p. 127-128. (ISBN 978-2-8041-0476-4).
Christian de Duve, Singularités : Jalons sur les Chemins de la Vie, Odile Jacob, Paris, Avril 2005, p.204-205. (ISBN 2-7381-1629-9)
(en) J. William Schopf 1994, Disparate rates, differing fates: Tempo and mode of evolution changed from the Precambrian to the Phanerozoic Proc. Natd. Acad. Sci. USA Vol. 91, pp. 6735-6742
(en) P. R. Yadav 2004, Prehistoric Life
(en) Gitai Z, « The new bacterial cell biology: moving parts and subcellular architecture », Cell, vol. 120, no 5, 2005, p. 577–86 (PMID 15766522, DOI 10.1016/j.cell.2005.02.026)
(en) Shih YL, Rothfield L, « The bacterial cytoskeleton », Microbiol. Mol. Biol. Rev., vol. 70, no 3, 2006, p. 729–54 (PMID 16959967, DOI 10.1128/MMBR.00017-06, lire en ligne)
(en) Norris V, den Blaauwen T, Cabin-Flaman A, et al, « Functional taxonomy of bacterial hyperstructures », Microbiol. Mol. Biol. Rev., vol. 71, no 1, mars 2007, p. 230–53 (PMID 17347523, PMCID 1847379, DOI 10.1128/MMBR.00035-06, lire en ligne)
(en) Nakabachi A, Yamashita A, Toh H, Ishikawa H, Dunbar H, Moran N, Hattori M (2006). The 160-kilobase genome of the bacterial endosymbiont Carsonella. Science 314 (5797): 267
(en) Pradella S, Hans A, Spröer C, Reichenbach H, Gerth K, Beyer S (2002). « Characterisation, genome size and genetic manipulation of the myxobacterium » Sorangium cellulosum So ce56. Arch Microbiol 178 (6): 484-92.
(en) Hinnebusch J, Tilly K (1993). « Linear plasmids and chromosomes in bacteria ». Mol Microbiol 10 (5): 917-22.
(en) Brüssow H, Canchaya C, Hardt W (2004). « Phages and the evolution of bacterial pathogens: from genomic rearrangements to lysogenic conversion ». Microbiol Mol Biol Rev 68 (3): 560-602.
(en) Perna N, Mayhew G, Pósfai G, Elliott S, Donnenberg M, Kaper J, Blattner F (1998). « Molecular evolution of a pathogenicity island from enterohemorrhagic Escherichia coli O157:H7 ». Infect Immun 66 (8): 3810-7.
(en) Denamur E, Matic I (2006). « Evolution of mutation rates in bacteria ». Mol Microbiol 60 (4): 820 – 7.
(en) Davison J (1999). « Genetic exchange between bacteria in the environment ». Plasmid 42 (2): 73 – 91.
(en) Hastings P, Rosenberg S, Slack A (2004). « Antibiotic-induced lateral transfer of antibiotic resistance ». Trends Microbiol 12 (9): 401
(en) Wu SC, Luo YM, Cheung KC, Wong MH. Influence of bacteria on Pb and Zn speciation, mobility and bioavailability in soil: a laboratory study. Environmental Pollution 2006; 144: 765‐773
(en) Nicholson W, Munakata N, Horneck G, Melosh H, Setlow P, « Resistance of Bacillus endospores to extreme terrestrial and extraterrestrial environments », Microbiol Mol Biol Rev, vol. 64, no 3, 2000, p. 548–72 (PMID 10974126, DOI 10.1128/MMBR.64.3.548-572.2000, lire en ligne)
(en) Siunov A, Nikitin D, Suzina N, Dmitriev V, Kuzmin N, Duda V, « Phylogenetic status of Anaerobacter polyendosporus, an anaerobic, polysporogenic bacterium », Int J Syst Bacteriol, vol. 49 Pt 3, 1999, p. 1119–24 (PMID 10425769, lire en ligne)
(en) Nicholson W, Fajardo-Cavazos P, Rebeil R, Slieman T, Riesenman P, Law J, Xue Y, « Bacterial endospores and their significance in stress resistance », Antonie Van Leeuwenhoek, vol. 81, nos 1–4, 2002, p. 27–32 (PMID 12448702, DOI 10.1023/A:1020561122764)
(en) Vreeland R, Rosenzweig W, Powers D, « Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal », Nature, vol. 407, no 6806, 2000, p. 897–900 (PMID 11057666, DOI 10.1038/35038060)
(en) Cano R, Borucki M, « Revival and identification of bacterial spores in 25- to 40-million-year-old Dominican amber », Science, vol. 268, no 5213, 1995, p. 1060–4 (PMID 7538699, DOI 10.1126/science.7538699)
(en) Nicholson W, Schuerger A, Setlow P, « The solar UV environment and bacterial spore UV resistance: considerations for Earth-to-Mars transport by natural processes and human spaceflight », Mutat Res, vol. 571, nos 1–2, 2005, p. 249–64 (PMID 15748651)
(en) Hatheway C, « Toxigenic clostridia », Clin Microbiol Rev, vol. 3, no 1, 1990, p. 66–98 (PMID 2404569, lire en ligne)
Voir aussi |
.mw-parser-output .autres-projets ul{margin:0;padding:0}.mw-parser-output .autres-projets li{list-style-type:none;list-style-image:none;margin:0.2em 0;text-indent:0;padding-left:24px;min-height:20px;text-align:left}.mw-parser-output .autres-projets .titre{text-align:center;margin:0.2em 0}.mw-parser-output .autres-projets li a{font-style:italic}
Bibliographie |
- Luciano Paolozzi, Jean-Claude Liébart, Microbiologie. Biologie des procaryotes et de leurs virus, Dunod, 2015, 514 p. (lire en ligne)
- (en) Larry L. Barton, Structural and functional relationships in prokaryotes, Springer Science & Business Media, 2005, 818 p. (lire en ligne)
Articles connexes |
- Bactéries photosynthétiques
- Bactéries chimiotrophes
- Structure bactérienne
- Procaryote phytopathogène
Liens externes |
Les procaryotes, Taxonomie et description des genres, Jean-Louis Garcia, Jean-Luc Cayol et Pierre Roger- J.P. Euzéby: List of Prokaryotic names with Standing in Nomenclature
- Portail de la microbiologie
- Portail de l’évolution